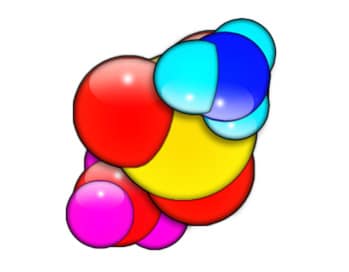
L’objectif du projet Folding@home, mené par l’université de Stanford, est d’aider la recherche scientifique en utilisant votre ordinateur et des milliers d’autres pour étudier le processus de repliement des protéines. Comme l’indique le Dr Vijay Pande, “Quand vous connaissez la forme d’une protéine, vous pouvez deviner ce qu’elle fait”. Si vous souhaitez aider la recherche, rejoignez sans plus tarder l’alliance francophone et l’équipe de Zebulon.fr !
Nous rejoindre
Pour rejoindre l’équipe Zebulon.fr de Folding@home et participer à cette expérience de calcul partagé nous vous conseillons de suivre les indications suivantes, cependant vous êtes libres de travailler de manière individuelle et vous pouvez participer sans rejoindre aucune équipe, l’important c’est de plier.
Téléchargement du client
Les clients Folding sont disponibles sur la page download du site officiel. Nous vous recommandons l’usage de la version console (vous pouvez quand même si vous le souhaitez utiliser la version graphique).
Installation du client
Le client console ne nécessite aucune installation particulière, il vous suffit de le copier dans un dossier de votre choix.
Configuration
Lors du premier lancement du client il vous faudra le configurer, pour figurer dans la mini-équipe Zebulon.fr sur les statistiques de l’alliance francophone il est obligatoire de respecter les règles suivantes:
Pseudo: [Zebulon.fr]_Votre pseudo
Numéro d’équipe: 51 ( C’est le numéro de l’alliance francophone)
Aide à la configuration
Si vous avez besoin d’aide lors de la configuration vous pouvez vous rendre sur le guide d’installation de la console.
Cacher la console
Vous pouvez utiliser le petit logiciel Fahtray qui permet de masquer ou de révéler la console de Folding en un clic.
Si vous êtes sous un système d’exploitation supportant la mise en service (Windows NT4, Windows 2000, Window XP) et que vous souhaitez de pas avoir à lancer le client à chaque démarrage, répondez yes lors de la configuration à la question Launch as a service?
Surveiller les pliages sous forme graphique
Vous pouvez utiliser le petit logiciel EMIII qui permet de voir différentes informations, telles que le temps moyen par step ou frame, la date et heure prévue de fin, le pourcentage en temps réel d’avancement de la step, de plus, on peut surveiller toutes les consoles lancées sur la machine ou même en réseau. Il suffit de lancer les consoles, puis le soft EMIII, après lui avoir indiqué les chemins des Consoles il reconnaîtra automatiquement si celles-ci sont lancées. Site officiel.
Bon Folding à tous 🙂
>> Rejoindre la discussion sur le forum <<
Le projet Scientifique
Nous allons essayer d’expliquer ce pourquoi vos PC moulinent comme des fous (la modélisation tridimensionnelle des protéines n’étant pas mon domaine particulier, je prie les puristes d’excuser les points de détail non abordées).
Les protéines sont des assemblages moléculaires très complexes, dont la fonction est fortement liée à leur conformation dans l’espace. Cette conformation est dépendante de nombreux facteurs physico-chimiques (température, pH, hydrophobicité), structuraux, et micro-environnementales (interaction protéines-protéines, transformation chimique sur des points clés de la structure)…
Une protéine se décrit selon 4 structures hiérarchisées:
-la structure primaire représente la séquence des acides aminés (a.a.=briques élémentaires) composant la protéine et peut aller de qq acides aminés à plusieurs centaines.
les acides aminés ont une structure commune organisée autour d’un atome de carbone. Ce carbone peut établir 4 liaisons chimiques fortes avec d’autres atomes (Carbone asymétrique sp4).
Ces 4 liaisons s’organisent à 120° l’une de l’autre, la structure d’un acide aminé est donc à la base tétraédrique, le carbone étant au centre du tétraèdre.
Structure commune des a.a. :
| H |
Les acides aminés s’emboîtent les uns dans les autres via la fonction acide (COOH) qui réagit avec la fonction amine (NH2) = liaison peptidique.
Donc déjà, première constatation, à chaque fois qu’on rencontre un C on tourne à 120° dans un plan.
Les acides aminés diffèrent entre eux par le radical R (qui peut être très court et peu encombrant=> un hydrogène pour la glycine; ou très long et ramifié pour d’autres acides aminés).
La nature physico-chimique de R (propriétés électrostatiques, hydrophobicité, encombrement stérique <= taille dans l’espace) impose des contraintes à la chaîne protéique.
En effet, les résidus hydrophobes dans un milieu hydrophile ont tendance à se regrouper ensemble, les résidus chargés positivement attire les résidus chargés négativement, les gros résidus imposent un angle particulier à la chaîne protéique, les acides aminés soufrés se relient entre eux par des ponts disulfure…
-la structure secondaire est dépendante des motifs basiques formés par certains enchaînement d’acides aminés.
En effet, on sait que si on retrouve une glycine (petit a.a) tous les 3 a.a suivie d’une proline (qui impose un coude à la chaîne protéique) et d’une deuxième série de glycine tous les 3 a.a, on obtiendra une structure en feuillet (dit beta).
D’autres structures secondaires existent, tels les hélices alpha qui forment une structure hélicoïdale avec des résidus hydrophobes à l’extérieur et des résidus hydrophiles à l’intérieur => ça va s’insérer dans les membranes lipidiques des cellules et créer des canaux (pour les ions par exemple).
-la structure tertiaire est le repliement de tout ça les uns sur les autres en prenant en compte toutes les forces d’attraction et de répulsion ainsi que d’encombrement stérique en fonction de l’environnement direct. La structure la plus stable (mais pas forcément la plus probable dans un milieu biologique) est celle qui a l’énergie potentielle la plus faible (on va pas rentrer dans la thermodynamique).
-finalement, la structure quaternaire représente les assemblages des sous-unités protéiques d’un gros complexe (ex: hémoglobine: 2 chaines de globine alpha + 2 chaines de globine beta, chaque globine portant un hème).
Pour compliquer le tout, les protéines peuvent subir des transformations au cours de leur maturation ou de leur vie au sein de la cellule (ajout de sucres, phosphorylation) qui vont directement influer sur leur structure 3D et donc sur leur fonction et les interactions qu’elles peuvent exercer avec leur environnement.
D’ailleurs, il existe des protéines dites “chaperonnes” qui aident la maturation d’une protéine à trouver sa forme dite normale et active.
Tous ces calculs doivent servir à déterminer les structures 3D les plus probables dans n conditions d’où la somme colossale de calculs.
Cela fera une base de connaissance mais cela restera que du calcul théorique (in silico). Quand on sait qu’une protéine isolée dans un tube à essai (in vitro) ne se comporte pas forcément (loin de là même) de la même façon que dans la cellule (in vivo), il reste du chemin à parcourir…
Gromacs et tinker sont apparemment des algorithmes de calcul utilisés dans la dynamique moléculaire. Mais c’est quoi donc la dynamique moléculaire ?
C’est une discipline à la croisée de la biologie moléculaire, les mathématiques, la physique et la chimie quantique qui étudie les trajectoires des atomes au cours du temps.
Pour vous faire voir un peu la complexité des calculs (qui dépendent du nombre d’atomes considéré) => ici
Une expérience de dynamique moléculaire se déroule en 3 étapes à partir des données structurales tridimensionnelles d’une molécule (ici les protéines) obtenues par diverses techniques telle la cristallographie par rayons X, la RMN 3D,…
1ère étape: thermalisation
on chauffe virtuellement la structure protéique pour casser les liaisons interatomiques faibles afin d’obtenir la séquence primaire.
2ème étape : équilibration
Stabilisation virtuelle de la molécule dépliée.
3ème étape : production
On suit à la femtoseconde près (10^-15 s = 1 millionième de milliardième de seconde) la trajectoire de tous les atomes constituant la protéine au cours de son repliement (repliement qui suit des lois thermodynamiques, physiques et chimiques précises)
Les atomes interagissant tous avec leurs voisins, la somme de calculs est relativement astronomique.
On peut imaginer qu’une step correspond au repliement d’une protéine à la femtoseconde donnée (ceci n’engage que moi et n’est pas le fruit d’une recherche poussée dans le fonctionnement du programme).
Les études de dynamique moléculaire se déroulait dans un vide virtuel, certaines interactions interatomiques n’étaient donc pas prises en compte. L’équipe de Stanford semble avoir intégré l’environnement aqueux à leur modélisation, ce qui collerait plus avec l’environnement cellulaire. (ce qui rajoute encore plus de calculs).
Une fois obtenue la cinétique précise de repliement des protéines, on peut envisager leur comparaison entre la protéine normale et celle identifiée dans les pathologies afin de déterminer à quel moment ça coince.
Tous ces calculs restent cependant très théorique car, malgré le fait de la prise en compte de l’environnement aqueux, je ne sais pas si l’intervention de protéines chaperonnes est prise en compte.
Voilà c’est à peu près tout ce dont je me souviens de mon post; si quelqu’un a des souvenirs un peu plus frais 😉
En espérant ne pas vous avoir trop barbé 😉
L’équipe de folding@home de Zebulon.fr
Courte introduction à l’utilisation de Folding@home par TheClem
Merci à DAVY pour l’idée et aux membre de 3Dchips-fr pour leur humour si indéfinissable …
Merci à Greywolf pour le relativement pas court texte explicatif
Merci à Folding@home pour nous donner l’impression de servir à quelque chose pauvres humains que nous sommes …
Pas merci au crash du disque dur qui nous a fait perdre 25 pages de ce topic.